Acoplamiento molecular de derivados de la 6-(piridin)-7H-indeno[2,1-c]quinolina como inhibidores de la Topoisomerasa 1 y PARP 1
DOI:
https://doi.org/10.24054/bistua.v20i2.1434Palabras clave:
interacciones molecularesResumen
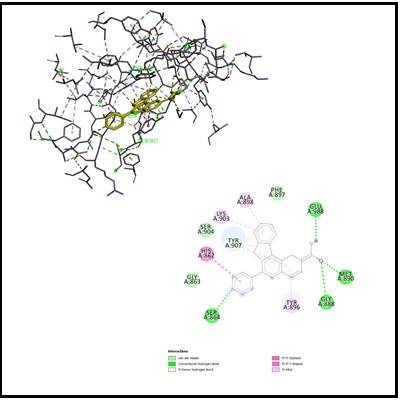
Una variedad de compuestos derivados de la 7H-indeno[2,1-c]quinolina han sido reportados ya que poseen propiedades anticáncer significativas. Sin embargo, los modos de acción aún no están claramente definidos. Se seleccionaron treinta derivados 6-(pyridin)-7H-indeno[2,1-c]quinolínicos los cuales exhiben actividad anticáncer y fueron sometidos a cálculos de acoplamiento molecular usando AutodockVina. Para investigar preliminarmente las dianas moleculares potenciales y confirmar la prueba de actividad experimental para estos compuestos anticancerígenos, el acoplamiento se realizó utilizando dos enzimas involucradas en el ciclo celular y en la replicación del ADN, e.g topoisomerasa 1 y PARP 1 (Poli [ADP-ribosa] polimerasa 1). Los resultados de acoplamiento revelaron que la 6-(piridin-4-il)-7H-indeno[2,1-c]quinolina-2-carboxamida 1j exhibió una mejor interacción de unión que el conocido inhibidor (D7N) co-cristalizado dentro de la (Poli [ADP-ribosa] polimerasa 1). Por otro lado, la 2-fluoro-6-(piridin-2-il)-7H-indeno[2,1-c]quinolina 3e se unió mejor a la ADN topoisomerasa 1. Los resultados obtenidos son útiles para comprender las características estructurales necesarias para mejorar las actividades inhibidoras.
Descargas
Citas
Li, J., Fu, A., & Zhang, L. (2019). An Overview of Scoring Functions Used for Protein–Ligand Interactions in Molecular Docking. Interdisciplinary Sciences – Computational Life Sciences, 11(2), 320–328. https://doi.org/10.1007/s12539-019-00327-w
Fu, Y., Zhao, J., & Chen, Z. (2018). Insights into the Molecular Mechanisms of Protein-Ligand Interactions by Molecular Docking and Molecular Dynamics Simulation: A Case of Oligopeptide Binding Protein. Computational and Mathematical Methods in Medicine, 2018. https://doi.org/10.1155/2018/3502514
Wu, Q., Peng, Z., Zhang, Y., & Yang, J. (2018). COACH-D: Improved protein-ligand binding sites prediction with refined ligand-binding poses through molecular docking. Nucleic Acids Research, 46(W1), W438–W442. https://doi.org/10.1093/nar/gky439
Ammar, Y. A., Sh El-Sharief, A. M., Belal, A., Abbas, S. Y., Mohamed, Y. A., Mehany, A. B. M., & Ragab, A. (2018). Design, synthesis, antiproliferative activity, molecular docking and cell cycle analysis of some novel (morpholinosulfonyl) isatins with potential EGFR inhibitory activity. European Journal of Medicinal Chemistry, 156, 918–932. https://doi.org/10.1016/j.ejmech.2018.06.061
AboulWafa, O. M., Daabees, H. M. G., & Badawi, W. A. (2020). 2-Anilinopyrimidine derivatives: Design, synthesis, in vitro anti-proliferative activity, EGFR and ARO inhibitory activity, cell cycle analysis and molecular docking study. Bioorganic Chemistry, 99(March), 103798. https://doi.org/10.1016/j.bioorg.2020.103798
Gurung, A. B., Ali, M. A., Bhattacharjee, A., Abul Farah, M., Al-Hemaid, F., Abou-Tarboush, F. M., Al-Anazi, K. M., Al-Anazi, F. S. M., & Lee, J. (2016). Molecular docking of the anticancer bioactive compound proceraside with macromolecules involved in the cell cycle and DNA replication. Genetics and Molecular Research, 15(2), 1–8. https://doi.org/10.4238/gmr.15027829
Fielden, J., Wiseman, K., Torrecilla, I., Li, S., Hume, S., Chiang, S. C., Ruggiano, A., Narayan Singh, A., Freire, R., Hassanieh, S., Domingo, E., Vendrell, I., Fischer, R., Kessler, B. M., Maughan, T. S., El-Khamisy, S. F., & Ramadan, K. (2020). TEX264 coordinates p97- and SPRTN-mediated resolution of topoisomerase 1-DNA adducts. Nature Communications, 11(1), 1–16. https://doi.org/10.1038/s41467-020-15000-w
Zhao, B., Liu, P., Fukumoto, T., Nacarelli, T., Fatkhutdinov, N., Wu, S., Lin, J., Aird, K. M., Tang, H. Y., Liu, Q., Speicher, D. W., & Zhang, R. (2020). Topoisomerase 1 cleavage complex enables pattern recognition and inflammation during senescence. Nature Communications, 11(1). https://doi.org/10.1038/s41467-020-14652-y
Takahashi, D. T., Gadelle, D., Agama, K., Kiselev, E., Zhang, H., Yab, E., Petrella, S., Forterre, P., Pommier, Y., & Mayer, C. (2022). Topoisomerase I (TOP1) dynamics: conformational transition from open to closed states. Nature Communications, 13(1). https://doi.org/10.1038/s41467-021-27686-7
Publicado
Cómo citar
Número
Sección
Licencia
Derechos de autor 2022 © Autores; Licencia Universidad de Pamplona.

Esta obra está bajo una licencia internacional Creative Commons Atribución-NoComercial-SinDerivadas 4.0.
© Autores; Licencia Universidad de Pamplona.





