Genetic diversity of Rubus glaucus Benth in the municipality of Pamplona (northeastern Colombia)
DOI:
https://doi.org/10.24054/bistua.v19i2.1125Keywords:
mora, PCR, diversidad genética, AMOVA, PICAbstract
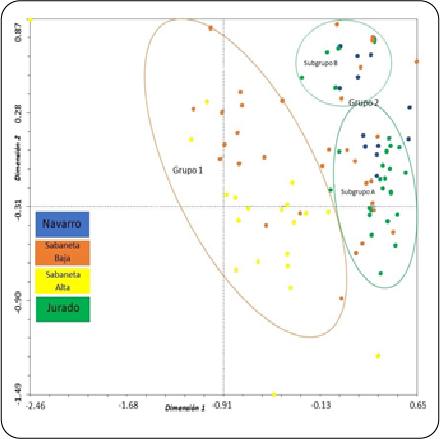
In Colombia, the area of R. glaucus Benth cultivation has increased since 2000, as it has organoleptic and economic characteristics of great attractiveness for national and international trade. The improvement of the supply of this product in the country, in a context of climate change, will depend on the implementation of new propagation techniques by farmers and the use of superior genotypes, with high diversity and free of pathogens. In this work, 7 polymorphic Simple Sequence Repeats (SSR) were selected to characterize the genetic fingerprint diversity in 92 individuals of R. glaucus in 23 farms of Pamplona (northeastern Andes of Colombia). DNA extraction was performed using the Invisorb® Spin plant kit. Microsatellites G01, R47a, R123a, R167h, R228a and R285a were amplified by PCR and revealed in 12% polyacrylamide gel. The DICE similarity index, analysis of molecular variation (AMOVA) and multiple correspondence analysis (MCA) reported that the blackberry population evaluated shows moderate to high genetic diversity (67% due to variation of individuals within farms and 33% between farms). The average values of polymorphic information content PIC = 0.302 and expected heterogeneity He = 0.388 indicate that the SSRs are suitable for future research on this species. This work contributes new data to the knowledge of genetic diversity in cultivars of R. glaucus in Colombia, and a set of microsatellites that provide useful information to implement better strategies for plant propagation of blackberry with the support of farmers in the region.
Downloads
References
D. Potter et al., “Phylogeny and classification of Rosaceae,” in Plant Systematics and Evolution, 2007, vol. 266, no. 1–2. doi: 10.1007/s00606-007-0539-9.
J. Y. Huang and J. M. Hu, “Revision of Rubus (Rosaceae) in Taiwan,” Taiwania, vol. 54, no. 4. 2009. doi: 10.6165/tai.2009.54(4).285.
Instituto de Ciencias Naturales, “Colección - hervario,” 2021. http://www.biovirtual.unal.edu.co/es/colecciones/result/genus/Rubus/plants/
J. Graham and M. Woodhead, “Raspberries and Blackberries: The Genomics of Rubus,” in Genetics and Genomics of Rosaceae, 2009. doi: 10.1007/978-0-387-77491-6_24.
M. Mimura, M. Mishima, M. Lascoux, and T. Yahara, “Range shift and introgression of the rear and leading populations in two ecologically distinct Rubusspecies,” BMC Evolutionary Biology, vol. 14, no. 1, p. 209, 2014, doi: 10.1186/s12862-014-0209-9.
Y. Wang et al., “Phylogenetic insight into subgenera Idaeobatus and Malachobatus (Rubus, Rosaceae) inferring from ISH analysis,” Molecular Cytogenetics, vol. 8, no. 1, 2015, doi: 10.1186/s13039-015-0114-y.
Q. Chen, Y. Wang, H. Nan, L. Zhang, H. Tang, and X. Wang, “Meiotic configuration and rDNA distribution patterns in Six Rubus taxa,” Indian Journal of Genetics and Plant Breeding, vol. 75, no. 2, 2015, doi: 10.5958/0975-6906.2015.00037.1.
K. Armes Carter, “Phylogenetic Estimation and Ancestral State Reconstruction of Rubus (Rosaceae) using Target Capture Sequencing,” Oregon State University, 2018.
A. M. López Gutiérez, M. L. Marulanda Ángel, L. M. Gómez López, and C. F. Barrera Sánchez, “Rubus glaucus Benth.: Morphology and floral biology aimed at plant breeding processes,” Revista Facultad Nacional de Agronomia Medellin, vol. 72, no. 3, 2019, doi: 10.15446/rfnam.v72n1.75910.
G. Franco and M. J. Giraldo, “El cultivo de la mora.” p. 83, 2001.
Additional Files
Published
Versions
- 2021-12-22 (5)
- 2022-03-23 (4)
- 2022-02-05 (3)
- 2021-12-23 (2)
- 2021-12-21 (1)
Issue
Section
License
Copyright (c) 2021 © Autores; Licencia Universidad de Pamplona.

This work is licensed under a Creative Commons Attribution-NonCommercial-NoDerivatives 4.0 International License.
© Autores; Licencia Universidad de Pamplona





